|
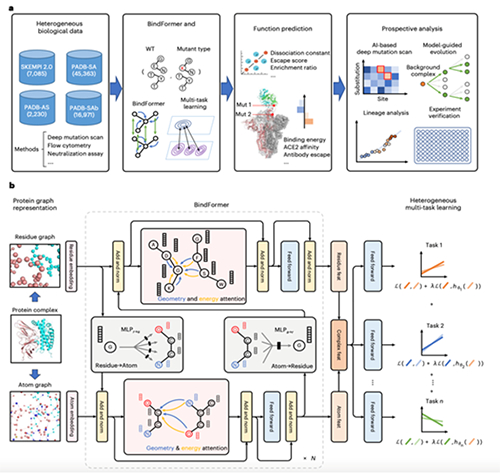 |
图 蛋白质功能分析框架UniBind
在国家自然科学基金项目(批准号:62272055)等资助下,北京邮电大学网络与交换技术全国重点实验室王光宇研究员,携手北京大学、澳门科技大学、伦敦大学学院等国内外多个生物医学团队开展交叉合作,在蛋白质功能分析、病毒进化趋势分析及AI 蛋白设计方面取得重要成果,实现了科学智能 (AI for Science, AI4S)领域研究的突破,通过“干湿结合”的科学研究闭环,加速或优化科学模拟和新发现。2023年7月31日在《自然•医学》(Nature Medicine)期刊上以“基于深度学习的蛋白质-蛋白质相互作用分析及预测SARS-CoV-2的传染性与变异进化(Deep-learning-enabled protein–protein interaction analysis for prediction of SARS-CoV-2 infectivity and variant evolution)”为题发表。论文链接:https://www.nature.com/articles/s41591-023-02483-5。
2021年DeepMind 发布的AlphaFold2实现了对蛋白质结构的准确预测,被视为“变革生命科学”的里程碑。然而,如何从蛋白质序列或结构出发,实现蛋白质复杂功能的预测,仍是一个重要挑战。蛋白质之间的相互作用(Protein-Protein Interaction, PPI)及亲和力(Affinity)是研究感染过程、免疫功能等生物学机理的关键。例如SARS-CoV-2病毒的刺突蛋白(S蛋白)与目标细胞上的血管紧张素转换酶2(ACE2)或中和抗体的相互作用,决定了病毒的受体亲和力和免疫逃逸能力。针对这一核心挑战,王光宇团队提出了一种通用的蛋白质功能分析框架UniBind(图),此框架包括涵盖残基和原子级别的多尺度图结构蛋白质表示方法、图结构蛋白特征提取的双路径神经网络BindFormer、以及BindFormer中的几何和能量注意力机制(GEA)模块。为了应对数据的多样性,团队采用多任务学习和模型集成方法训练UniBind,从多源异构的生物数据集中提取关联信息,以增强其预测的鲁棒性。
王光宇研究团队使用SKEMPI和SKEMPI v.2.0等主要基准数据集进行系统测试和验证,结果表明UniBind具有准确性、鲁棒性和可扩展性。通过分析全球流感数据库中的六百多万病毒序列,UniBind可预测何种突变会导致病毒的传染力增加,或使病毒对抗体或疫苗产生抗性。团队利用UniBind模拟了三万多株已知的病毒毒株,并正确预测了目前占主导地位突变株的演变。此外,基于可溶性ACE2可作为诱饵蛋白中和SARS-CoV-2感染这一事实,UniBind采用了计算机模拟演化的方法,筛选了一系列候选蛋白,并联合生物合作团队开展“干湿结合”实验,验证了AI设计的高亲和力ACE2受体诱饵分子具有防治当前和未来病毒变体的广泛潜力。此工作为蛋白质与蛋白质复合物的亲和力分析提供了一个通用计算框架,并为未来疫情的早期监控及加速疫苗研发奠定了基础。